用戶文章:結合質譜蛋白質測序解碼蛋白質糖基化
糖蛋白占人類蛋白質和大多數生物制藥產物的50%以上,蛋白質糖基化作為最廣泛和最復雜的翻譯后修飾(PTM)之一,對調節各種生物過程至關重要。糖蛋白序列的綜合分析,包括糖基化位點和相關聚糖結構的深度剖析,是糖蛋白功能研究的關鍵。液相色譜-質譜法(LC-MS/MS)已成為蛋白質鑒定和翻譯后修飾發現的強大工具。與糖蛋白質組學中廣泛使用的數據庫搜索的策略不同,蛋白質從頭測序無需事先了解DNA/氨基酸序列,用于未知蛋白的序列測定。然而,多糖基化位點的復雜性通常會使得質譜很難獲得信息豐富的糖肽碎片離子,導致序列覆蓋不完整和游離寡糖修飾譜不明確,從而影響從頭測序的準確性。
2025年初,上海藥物研究所的周虎研究員和文留青研究員聯合在Analytical Chemistry期刊聯合發表了一種結合質譜蛋白質從頭測序技術解碼未知糖蛋白的新方法:Decoding Protein Glycosylation by an Integrative Mass Spectrometry-Based De Novo Sequencing Strategy,方案設計見圖1。Fetuin-A(UniProt Accession no.P12763)是一種含有唾液酸化N糖和O糖的常用模型糖蛋白,作者首先基于Fetuin-A,用糖苷酶切除糖鏈后采集質譜數據,使用PEAKS® AB完成蛋白質序列的自動組裝,建立了糖蛋白的從頭測序方法,可獲得去糖基化蛋白的一級序列。接下來,使用高度復雜的糖基化蛋白藥物依那西普來驗證該方法的性能。隨后,作者應用該方法鑒定了三種未知 TNFR:Fc 融合生物制劑的氨基酸序列,以探索它們與原研藥物依那西普的相似性。并且,從N糖基化位點、游離糖、糖蛋白亞單位、糖肽的多個層面進行了深度糖基化表征,以精確定位 N−/ O-糖基化。該方法彌合了從頭測序和糖基化修飾分析之間的差距,提供了有關糖蛋白一級結構和糖基化修飾的全面信息,在基礎研究和生物制藥行業均具有實用價值。
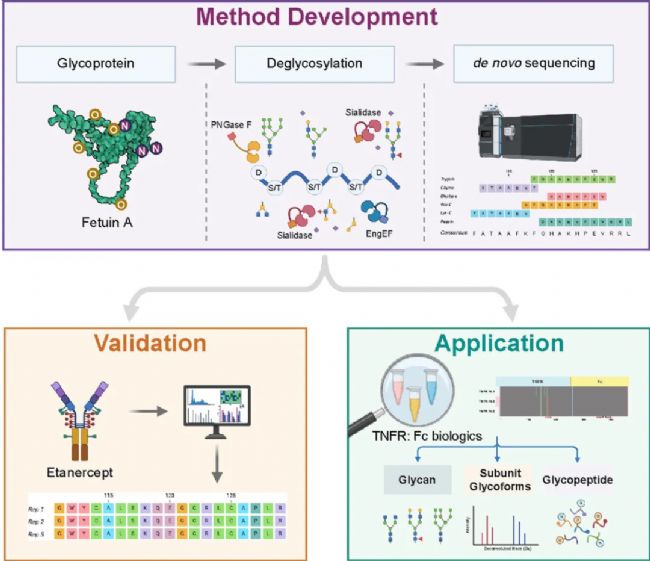
圖1 方案設計示意圖
方法與結果
糖蛋白從頭測序方法開發
使用PNGase F和O-糖苷酶 EngEF 分別去除Fetuin A上的 N糖和O糖,然后通過Intact Mass驗證糖基化切除效果。去除聚糖后,大多數酶切的序列覆蓋率顯著提高,chymotrypsin、Glu-C、pepsin和trypsin的序列覆蓋率均可達到90% 以上。考慮到不同酶切位點的互補性,后續將LysC、Elestase、chymotrypsin、Glu-C、pepsin和trypsin6種酶結合使用,結果如預期,去糖后的序列可信度進一步提升。去除糖基后,糖肽骨架的鑒定更可靠,PEAKS® AB可直觀展示切糖前后的序列覆蓋情況(圖2)。
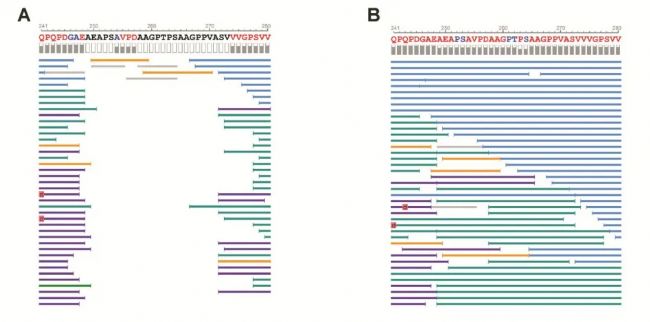 圖2 PEAKS® AB序列覆蓋圖
圖2 PEAKS® AB序列覆蓋圖獲得足夠豐富的多肽碎片離子對于揭示蛋白質相鄰氨基酸的序列至關重要。EThcD碎裂可顯著增加MS/MS譜圖中豐富的碎片離子,有利于多肽序列和翻譯后修飾的鑒定,因此作者評估了EThcD碎裂方法與常用的階梯式HCD碎裂方法。結果表明,盡管de novo候選肽的總數少于HCD 方法,但 EThcd在肽序列長度(圖 3B)和譜圖質量(圖 3C)方面更優,EThcD譜中較長的多肽對于蛋白質序列的組裝有利(圖3D-E)。更重要的是,考慮到測序準確度,EThcD方法的平均序列準確度高達98.3%的,而HCD的平均準確度為95.8%且包含1個gap(圖3F)。
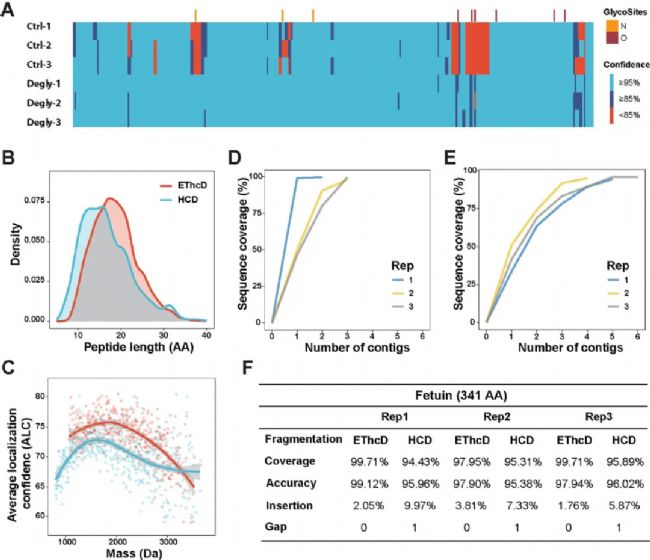
圖3 Fetuin A蛋白測序實驗方法比較
蛋白質測序中,Ile-Leu(I-L)和Asn-Asp(N-D)的確證往往需要更多的譜圖信息。PNGase F切除N糖后,會將糖基化Asn殘基轉化為Asp,EThcD和HCD均可較好的鑒定出來。N-糖基化的Asn殘基可以通過18O標記的肽圖分析法以及基于糖苷酶的方法進行確定。值得注意的是,EThcD 方法在Leu/Ile測定方面具備獨特的優勢,利用EThcD譜圖中w離子與z離子的質量差(Leu,-43.05 Da;Ile,-29.04 Da),可明確鑒定蛋白序列中的I和L(例如肽 DIEIDTLETTCH,圖4)。
綜合以上結果,作者明確了蛋白質測序的基本實驗方法,即切糖與EThCD采集方法結合。
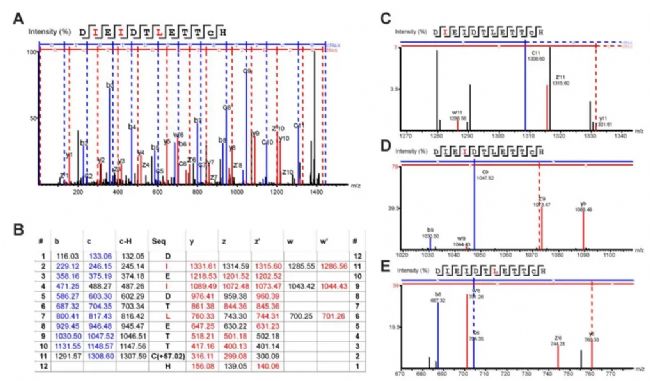 圖4 EThcD示例肽段
圖4 EThcD示例肽段依那西普從頭測序
為了測試前述蛋白質從頭測序方法的穩健性并證明在高度糖基化生物制藥中的應用效果,作者對已上市的治療性Fc融合蛋白原藥依那西普進行從頭測序。作為最復雜的高度糖基化Fc 融合生物藥物之一,依那西普具有至少3個N-和13個O-糖基化位點。對依那西普切除糖基化后,通過 EThcD 碎裂方法獲得MS/MS譜圖,用PEAKS® AB完成蛋白質自動測序。三個重復樣本的測序結果的覆蓋度均達到98.93%,準確度為99.57-100%(表 1)。同時,不切糖的常規策略無法完成序列組裝,可能是因為高度糖基化導致MS/MS譜過于復雜,形成的序列gap較多,無法通過從頭測序算法和多酶酶切的方法解決(圖 5)。且未覆蓋的區域位于鉸鏈區,這在人類蛋白質組數據庫中是不存在的,這表明使用標準數據庫搜索方法推斷高度糖基化蛋白的全長一級序列具有挑戰性。總之,本研究方法能夠對糖蛋白進行從頭測序,并實現糖基化修飾區域的深度覆蓋。
表1 依那西普切糖測序重復性
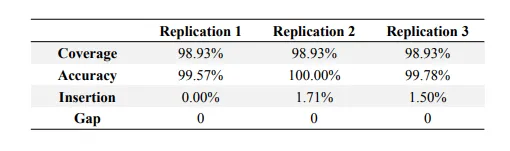
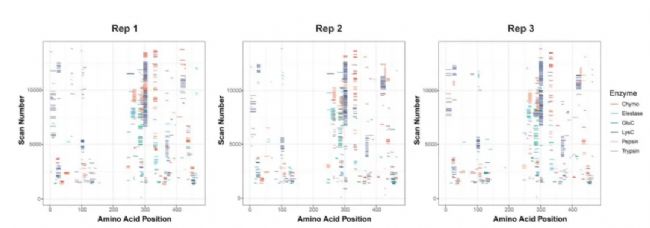 圖5 不切糖的依那西普多酶酶切
圖5 不切糖的依那西普多酶酶切未知TNFR:Fc融合生物制劑的從頭測序
應用上述方法對三種依那西普的生物仿制藥TNFR:Fc融合生物制劑進行測序。Intact Mass結果顯示,TNFR:Fc 2和TNFR:Fc 3與依那西普存在細微差異,而TNFR:Fc 1的完整質量與依那西普相同。與之對應,這三個生物制劑的測序結果顯示,有三個氨基酸存在差異,即M174R(在TNFR片段中), E376D 和M378L(均在 Fc片段中)(圖 6A),對應于變異位點的 MS/MS譜圖碎片離子質量均較高(圖 6B-E)。此外,蛋白質序列的差異與完整的蛋白質質量一致(圖 6F-G)。M174R上的序列變異是人TNFR 2編碼區的多態性導致的,根據報道,應該與多囊卵巢綜合征、高雄激素血癥和系統性紅斑狼瘡有關。同樣,在市售的TNF 2受體-Fc 融合蛋白產品中也報道了Fc片段的E376D和M378L突變。我們使用 PEAKS® AB軟件的Sequence Validation工作流驗證了三個TNFR:Fc融合生物制劑蛋白序列。∼99%的序列的可信度>85%,且每條肽段均有多張譜圖支持。因此,該結果證實了從頭測序結果的高精度。
應用上述方法對三種依那西普的生物仿制藥TNFR:Fc融合生物制劑進行測序。Intact Mass結果顯示,TNFR:Fc 2和TNFR:Fc 3與依那西普存在細微差異,而TNFR:Fc 1的完整質量與依那西普相同。與之對應,這三個生物制劑的測序結果顯示,有三個氨基酸存在差異,即M174R(在TNFR片段中), E376D 和M378L(均在 Fc片段中)(圖 6A),對應于變異位點的 MS/MS譜圖碎片離子質量均較高(圖 6B-E)。此外,蛋白質序列的差異與完整的蛋白質質量一致(圖 6F-G)。M174R上的序列變異是人TNFR 2編碼區的多態性導致的,根據報道,應該與多囊卵巢綜合征、高雄激素血癥和系統性紅斑狼瘡有關。同樣,在市售的TNF 2受體-Fc 融合蛋白產品中也報道了Fc片段的E376D和M378L突變。我們使用 PEAKS® AB軟件的Sequence Validation工作流驗證了三個TNFR:Fc融合生物制劑蛋白序列。∼99%的序列的可信度>85%,且每條肽段均有多張譜圖支持。因此,該結果證實了從頭測序結果的高精度。
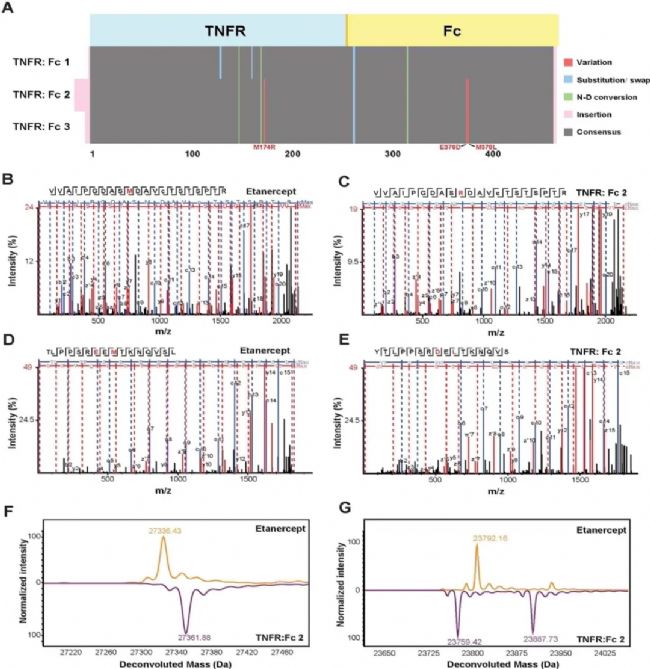 圖6 未知TNFR:Fc融合蛋白測序
圖6 未知TNFR:Fc融合蛋白測序TNFR:Fc融合生物制劑的N-/O-糖基化分析
作者從N-糖基化位點鑒定、游離糖、糖蛋白亞基和糖肽多個水平對TNFR:Fc融合制劑進行了全面的N和O糖基化剖析。首先通過18O標記對N糖基化位點進行定量(圖7A)。通過計算18O位點所在肽段的強度,在四個TNFR:Fc 融合蛋白樣品中篩選出三個N-糖基化位點(N149、N171 和 N317)(圖 7B),這三個N糖基化位點與依那西普中已知的N糖基化位點完全匹配。此外,作者通過內切糖苷酶混合物(Endo CC、Endo S 和 Endo H)部分裂解 N糖,產生帶有"GlcNAc"/"GlcNAc−Fuc" 的N位點碎片,以確認N-糖基化位點的鑒定結果(圖 7A、C)。使用酶切特異性更廣的糖苷酶混合物,對N糖鏈進行充分切割,結果與18O標記高度一致(圖 7D)。這些結果均可用于驗證從頭測序結果中的Asn或Asp。
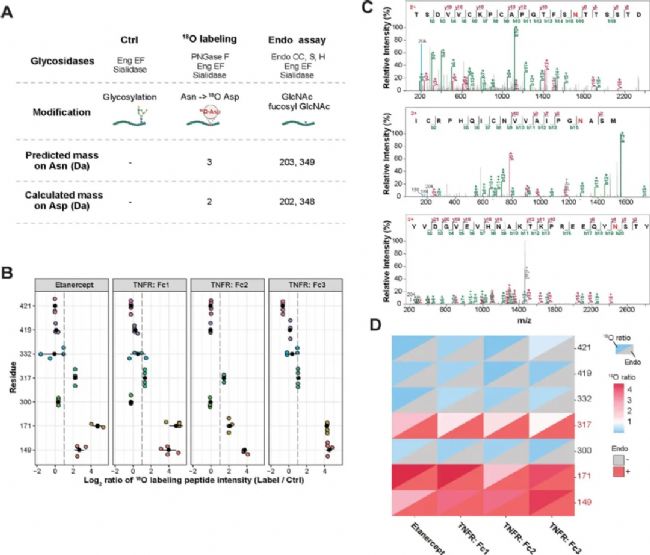 圖7 N糖基化位點鑒定
圖7 N糖基化位點鑒定為了在整個蛋白質水平上確定N糖的含量和結構,作者又用2-AB標記對酶促釋放的N糖進行衍生化,然后通過UPLC-HILIC-FLR-MS 進行熒光和質譜檢測。依那西普釋放的N糖中,最豐富的峰是 F(6)G2F(雙天線,核心巖藻糖基化,具有兩個末端半乳糖),其次是 F(6)G0F(雙天線,核心巖藻糖基化,無末端半乳糖殘基),G2(雙天線,具有兩個末端半乳糖殘基),以及 F(6)G1F(雙天線,核巖藻糖基化,帶有一個末端半乳糖殘基)(圖 8A)。三種TNFR:Fc融合生物制劑的結果與依那西普相似。然后,作者去除O糖和唾液酸,在亞基水平上評估了N糖的不同糖型。依那西普/TNFR:Fc 1的TNFR片段處豐度最高的糖型為G2 和 G2F(圖 8B)。而TNFR:Fc 3的TNFR片段除了主要糖型G2和G2F外,還包含許多G1F(圖 8C)。另一方面,盡管觀察到微小程度的賴氨酸丟失修飾,但在四個樣品中,Fc片段處最突出的糖型均為G0F和G1F。
為了降低糖肽的復雜性和可變性,提高糖肽的酶切效率,作者將唾液酸酶與N-糖苷酶或O糖苷酶結合使用,以深入了解位點特異性 N-/O-糖基化。結果如圖8D,即N149、N171 和 N317 糖基化位點主要被G2、G2F 和G0F占據。N171是導致TNFR:Fc 3糖型差異的位點,含有大量的G1F。位點特異性N-糖肽結果與基于亞基水平的糖型分配一致,驗證了不同酶切水平下結果的一致性。
結合手動校驗糖肽譜圖,作者鑒定出了依那西普和3個TNFR:Fc 融合生物制劑上的13個O-糖基化位點(圖 8E)。與之前的報道的12個O-糖基化位點一致。在TNFR:Fc融合生物制劑中觀察到類似的糖基化模式。此外,對糖基化位點的分析揭示了鉸鏈區的高度糖基化(圖 8F)。盡管依那西普和 TNFR:Fc 融合生物制劑在骨架上存在結構相似,但由于不同的制造工藝和細胞來源,它們在糖譜上表現出差異。這些發現突出了潛在的物理化學意義,值得進一步研究,并有助于依那西普及其生物仿制藥的全面表征。
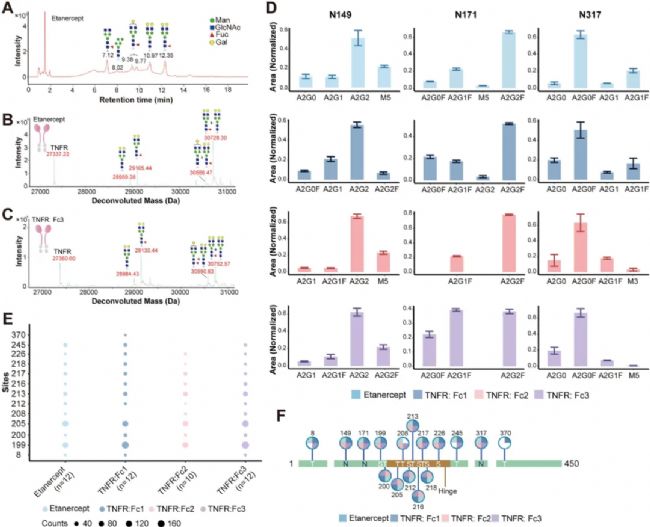 圖8 依那西普與融合制劑的糖型表征
圖8 依那西普與融合制劑的糖型表征原文鏈接:https://pubs.acs.org/doi/10.1021/jacsau.4c00960?ref=PDF
若您想深入了解PEAKS® AB的功能和應用,可點擊“閱讀原文”提交您的咨詢信息。

-掃碼關注-
www.bioinfor.com (EN)
www.deepproteomics.cn(CN)
作為生物信息學的領軍企業,BSI專注于蛋白質組學和生物藥領域,通過機器學習和先進算法提供世界領先的質譜數據分析軟件和蛋白質組學服務解決方案,以推進生物學研究和藥物發現。我們通過基于AI的計算方案,為您提供對蛋白質組學、基因組學和醫學的卓越洞見。旗下著名的PEAKS®️系列軟件在全世界擁有數千家學術和工業用戶,包括:PEAKS®️ Studio,PEAKS®️ Online,PEAKS®️ GlycanFinder, PEAKS®️ AB,DeepImmu®️ 免疫肽組發現服務和抗體綜合表征服務等。
聯系方式:021-60919891;sales-china@bioinfor.com
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com